定义距离和相似性

距离的直观定义
pacman::p_load(tidyverse,ggthemes,ggfortify,DT)
two_person <- tribble(~x, ~y,
0,0,
9,12)
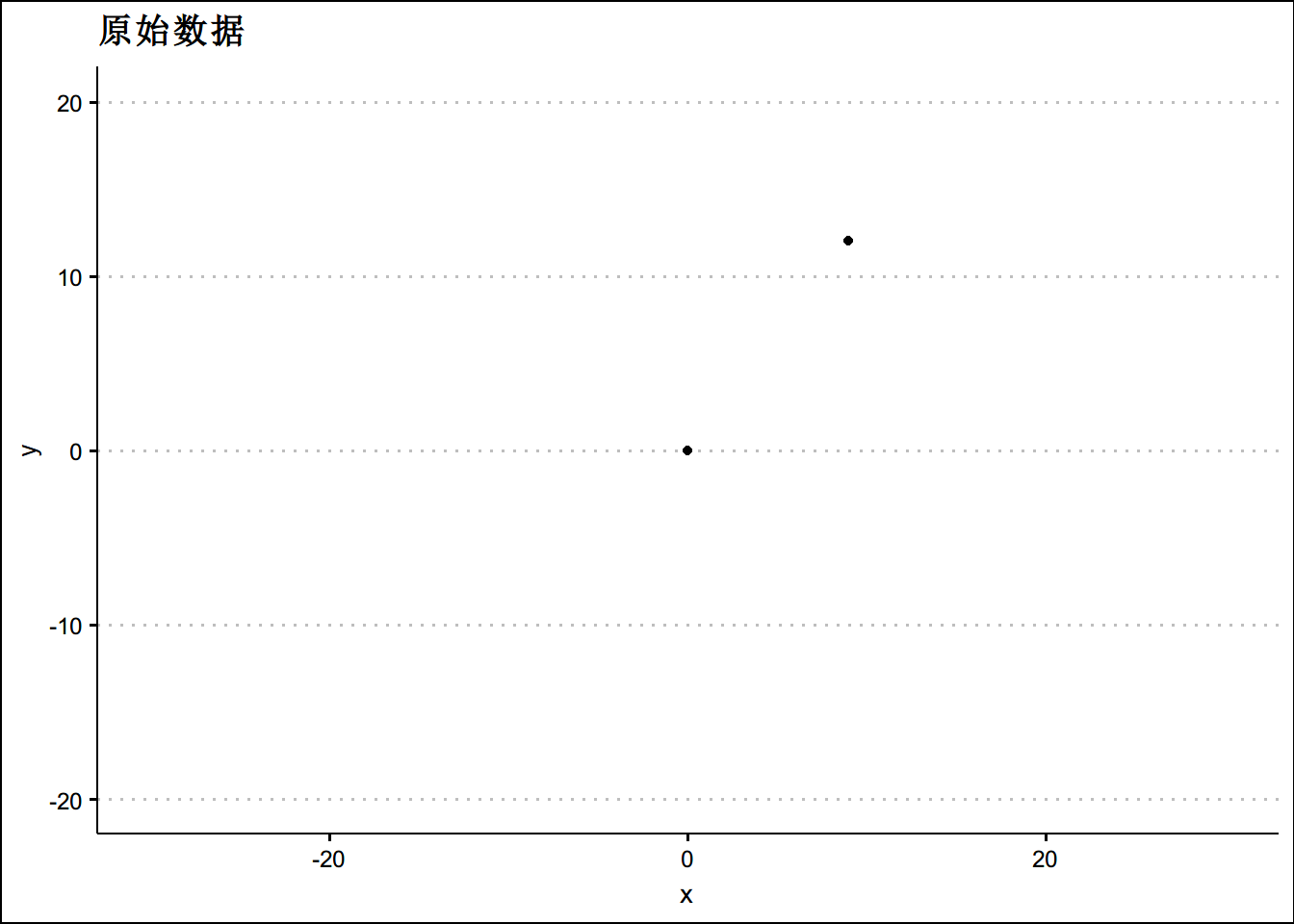
ggplot(two_person, aes(x = x, y = y)) +
geom_point() +
lims(x = c(-30,30), y = c(-20, 20)) + theme_clean()+ggtitle("原始数据")dist(two_person,method = "euclidean")## 1
## 2 15多余两个人时
three_p <- tribble(~x, ~y,
0,0,
9,12,
-2,9)
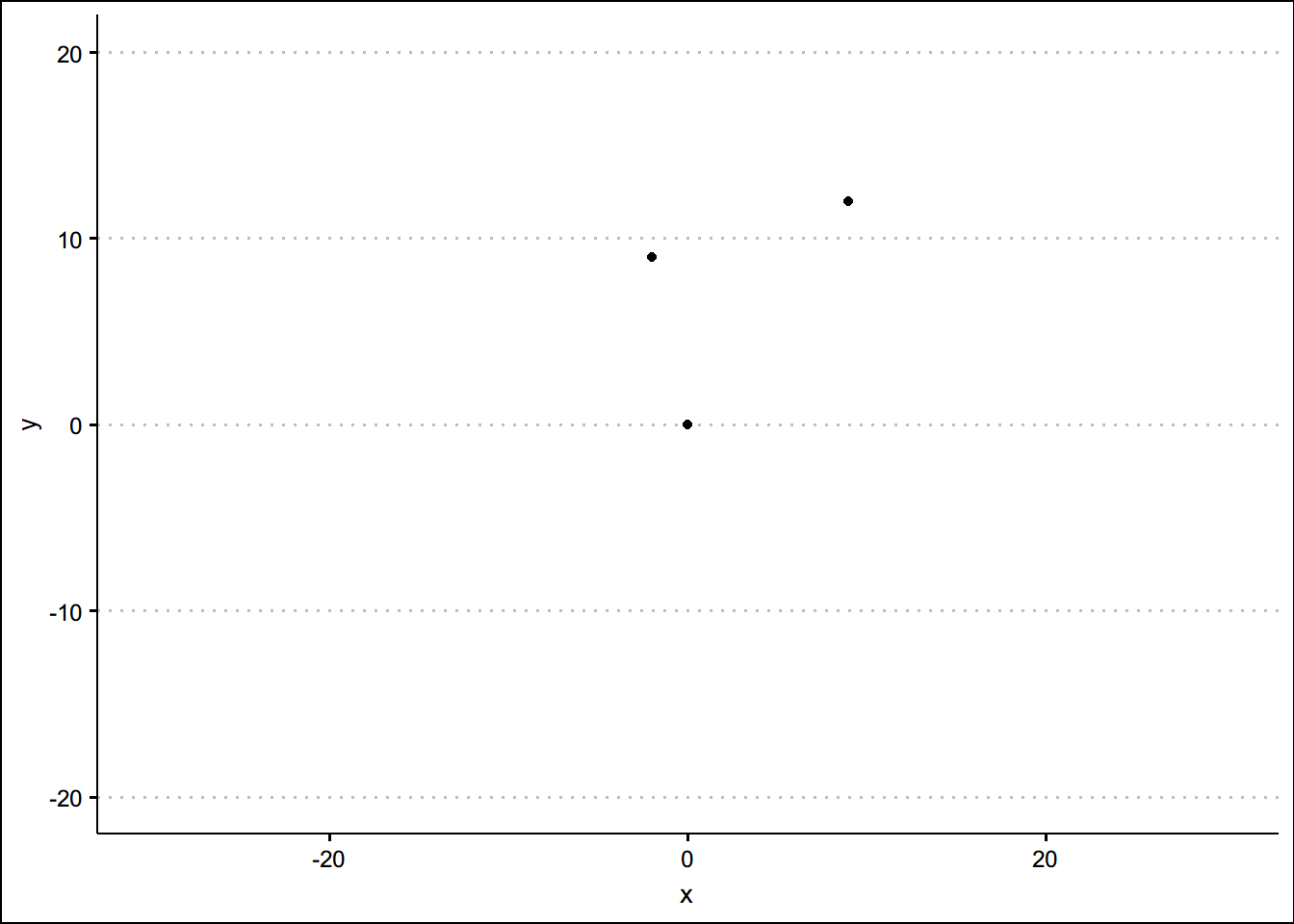
ggplot(three_p, aes(x = x, y = y)) +
geom_point() +
lims(x = c(-30,30), y = c(-20, 20)) + theme_clean()dist(three_p,method = "euclidean")## 1 2
## 2 15.000000
## 3 9.219544 11.401754大家思考一下 1和2,2和3,1和3哪个离得最近(相近性)最高?
数据尺度(scale)的影响
我们在计算样本特征的相似度时,需要注意数据尺度的影响
three_person <- tribble(~weight,~height,
8.3,840,
8.6,780,
10.5,864)
three_person %>% datatable()dist(three_person)## 1 2
## 2 60.00075
## 3 24.10062 84.02149scale(three_person)## weight height
## [1,] -0.6984984 0.2773501
## [2,] -0.4470390 -1.1094004
## [3,] 1.1455375 0.8320503
## attr(,"scaled:center")
## weight height
## 9.133333 828.000000
## attr(,"scaled:scale")
## weight height
## 1.193035 43.266615dist(scale(three_person))## 1 2
## 2 1.409365
## 3 1.925659 2.511082上面一个简单的例子,我们可以看出数据的尺度对我们计算距离有很大影响。
如何计算类别变量(categorical)的距离
我们还是看一个例子
kouwei <- tribble(~la,~suan,~tian,~chou,
T,T,F,F,
F,T,T,T,
T,T,F,T)
kouwei %>% datatable()如何计算上述口味例子的距离呢?
- 首先我们定义 来计算类别型变量的相似度
- 假设我们希望计算样本1和样本2的距离,根据上面的定义,我们有 那么1和2的距离为 如果使用软件计算
dist(kouwei,method = 'binary')## 1 2
## 2 0.7500000
## 3 0.3333333 0.5000000如果类别变量多于两个,如何计算距离与相似度?如果你的数据如下,如何处理?
teacher_sat <- tribble(~taidu,~fangfa,~renpin,
'high','low','high',
'low','high','mid',
'mid','mid','mid')
teacher_sat %>% datatable()面对上述类型的数据,我们需要将多种类型数据处理成虚拟变量,才能进行距离和相似度的计算
library(dummies)
teacher_sat_df <- as.data.frame(teacher_sat)
teacher_sat_dum <- dummy.data.frame(teacher_sat_df)
teacher_sat_dum## taiduhigh taidulow taidumid fangfahigh fangfalow fangfamid renpinhigh
## 1 1 0 0 0 1 0 1
## 2 0 1 0 1 0 0 0
## 3 0 0 1 0 0 1 0
## renpinmid
## 1 0
## 2 1
## 3 1dist(teacher_sat_dum,method = 'binary')## 1 2
## 2 1.0
## 3 1.0 0.8teacher_sat## # A tibble: 3 x 3
## taidu fangfa renpin
## <chr> <chr> <chr>
## 1 high low high
## 2 low high mid
## 3 mid mid mid如果我们有如下距离矩阵,请思考以下问题:
| 1 | 2 | 3 | |
|---|---|---|---|
| 2 | 11.7 | ||
| 3 | 16.8 | 18 | |
| 4 | 10 | 20.6 | 15.8 |
首先可以肯定的是我们知道样本1和样本4的距离最近
样本2离group 1,4更近?还是样本3离group 1,4更近?
如何判断上述问题的距离?
一种想法:,,那么3离group1,4比2更近一些。
图解层次聚类








接下来我们扩展一下这个思路,再多放几个队员上场,假设我们现在有6个队员,他们的x坐标和y轴坐标为:
| player | x | y |
|---|---|---|
| 1 | -1 | 1 |
| 2 | -2 | -3 |
| 3 | 8 | 6 |
| 4 | 7 | -8 |
| 5 | -12 | 8 |
| 6 | -15 | 0 |
根据以上数据,我们对上面6个球员进行层次聚类Hierarchical clustering
player_6 <- tribble(~x,~y,
-1,1,
-2,-3,
8,6,
7,-8,
-12,8,
-15,0
)
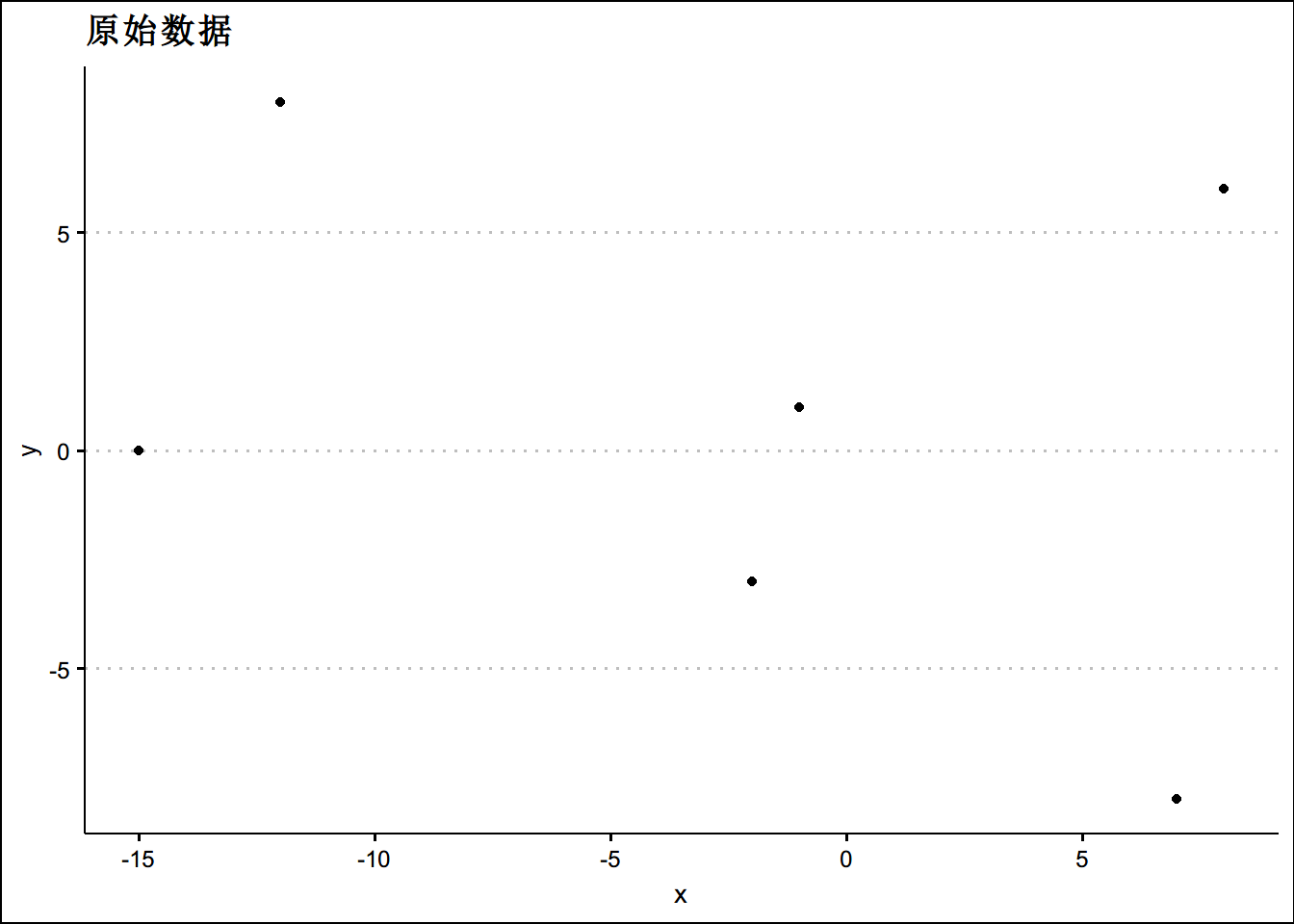
# 看一眼球员的位置
ggplot(player_6,aes(x=x,y=y))+geom_point()+theme_clean()+ggtitle("原始数据")dist_players <- dist(player_6, method = 'euclidean')
hc_players <- hclust(dist_players, method = 'complete')
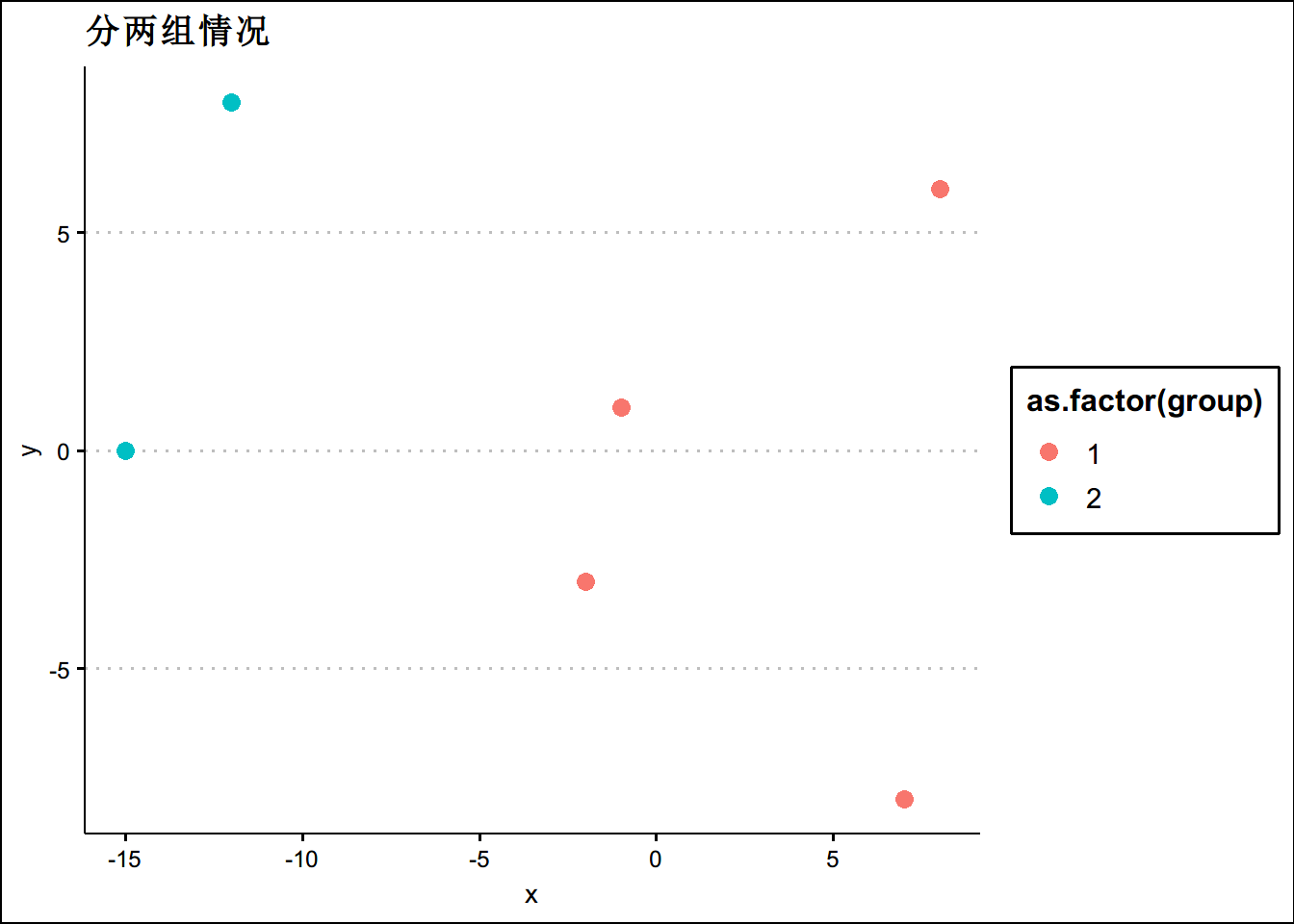
#如果我们要将数据分成两组,用cuttree剪树枝,这里我手画一个图给大家理解
cluster_assignments <- cutree(hc_players, k = 2)
# 把2组分类的标准装回数据框
play_6_k2 <- player_6 %>%
mutate(group = cluster_assignments)
ggplot(data = play_6_k2 ,aes(x=x,y=y,color=as.factor(group)))+geom_point(size=3)+theme_clean()+
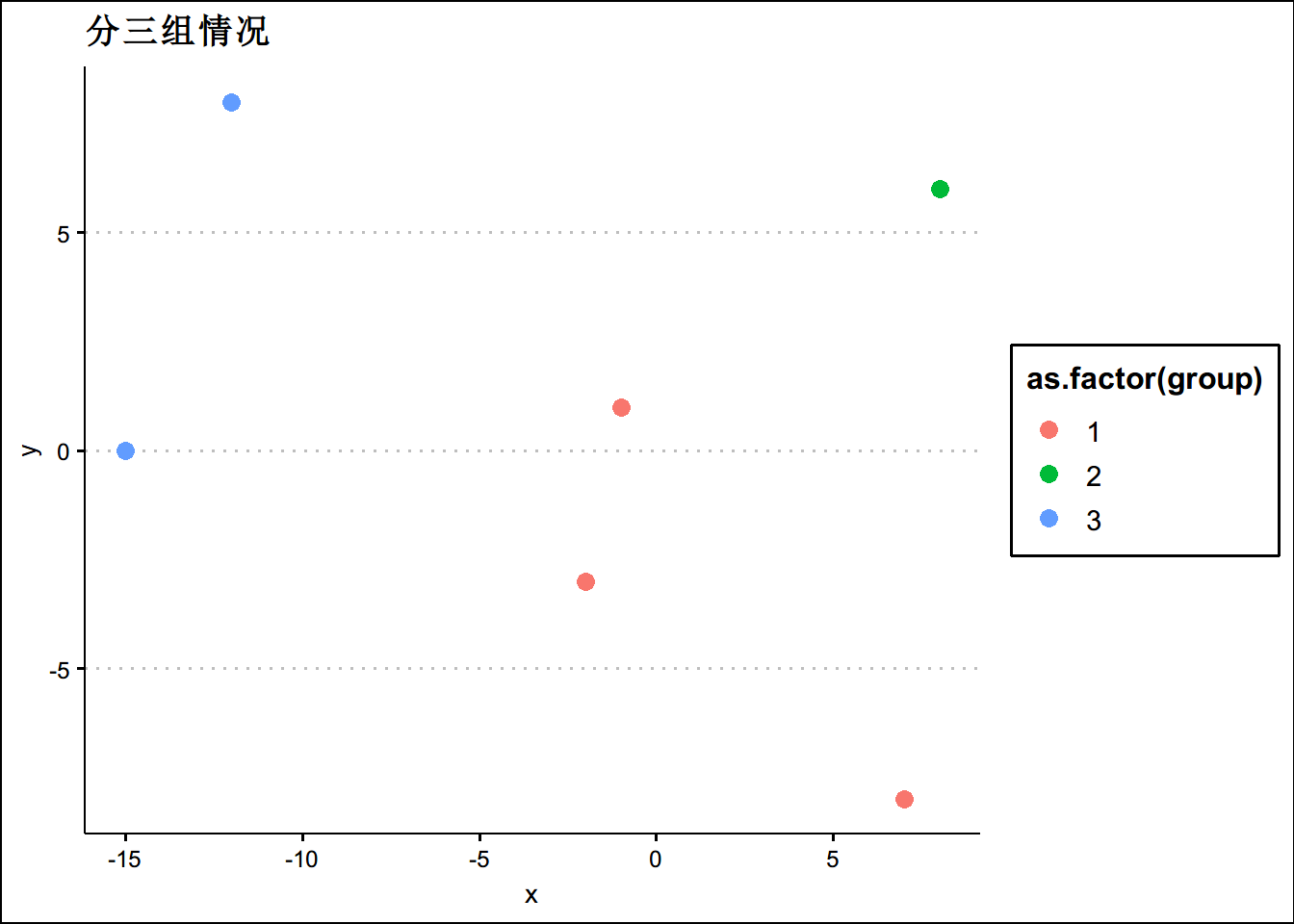
ggtitle('分两组情况')cluster_assignments <- cutree(hc_players, k = 3)
# 把2组分类的标准装回数据框
play_6_k2 <- player_6 %>%
mutate(group = cluster_assignments)
ggplot(data = play_6_k2 ,aes(x=x,y=y,color=as.factor(group)))+geom_point(size=3)+theme_clean()+
ggtitle('分三组情况')Kmeans的思想:
一个好的聚类算法,应该是能够使得类内的差异尽可能小。设是第类中差异化的度量,因此我们的问题转化为:
使用平方欧式距离定界定类内差异
具体算法:
1 随机为每个观测值分配一个1到K的数字
2 遍历所有数据,将每个数据划分到最近的中心点中
3 计算每个聚类的平均值,并作为新的中心点
4 重复2-3,直到这k个中线点不再变化(收敛了),或执行了足够多的迭代
上图展示了K=3的Kmeans聚类算法过程,先给每个观测值随机分配1-3的数字,然后算出这些1-3类的中心,第一行第三个图展示了这个中心;左下第一张图,每个观测值被分配到了与之最接近的类中;第二行第二列图,重新计算不同类的中心;第二行第三列反复迭代后的结果。我们先虚拟一个项目
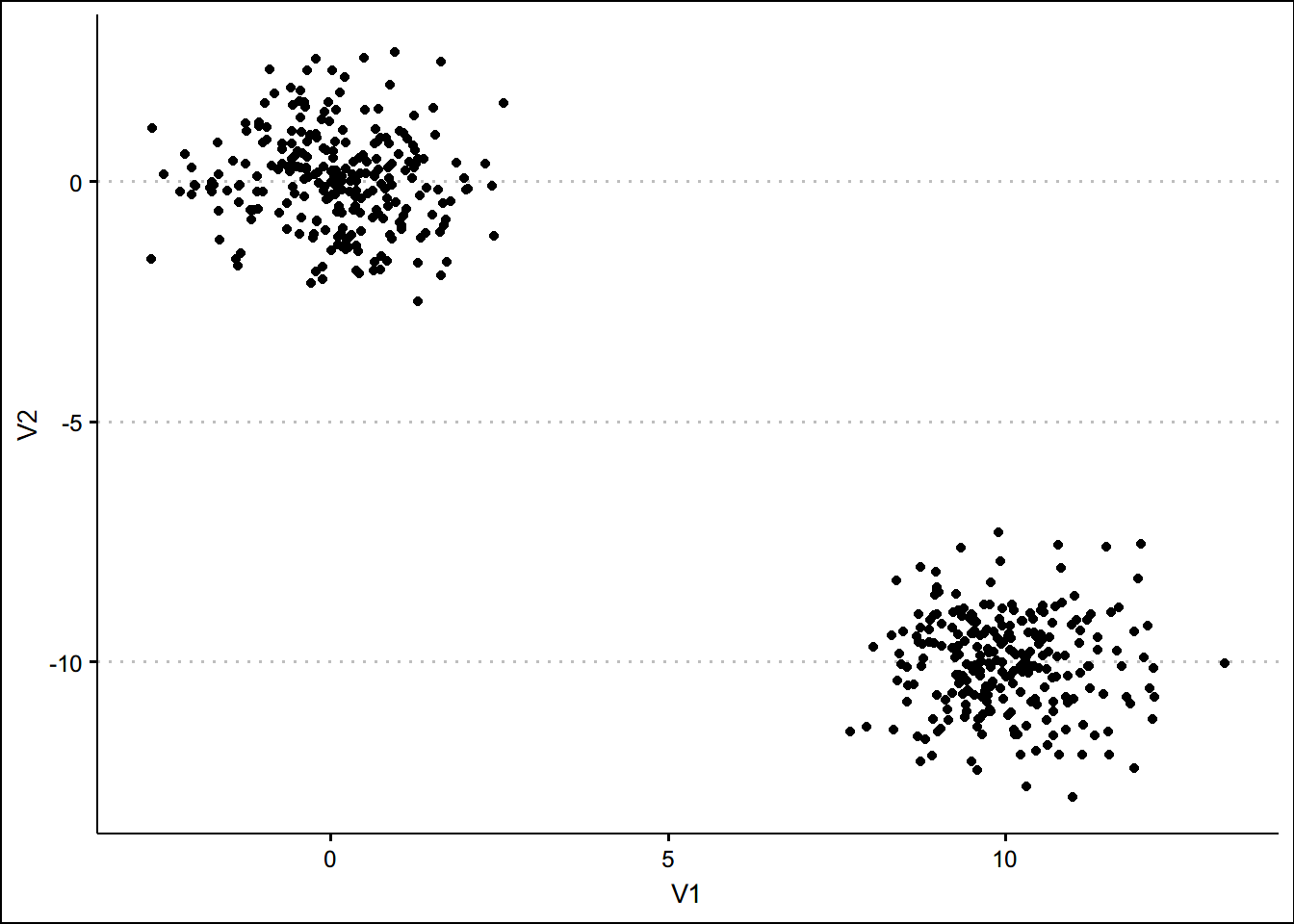
set.seed(123)
x <- matrix(rnorm(500*2),ncol=2)
x[1:250,1] <- x[1:250,1]+10 #分别加10和减去10
x[1:250,2] <- x[1:250,2]-10
x <- as.tibble(x)
ggplot(data = x ,aes(x=V1,y=V2))+geom_point()+theme_clean()#看一眼数据
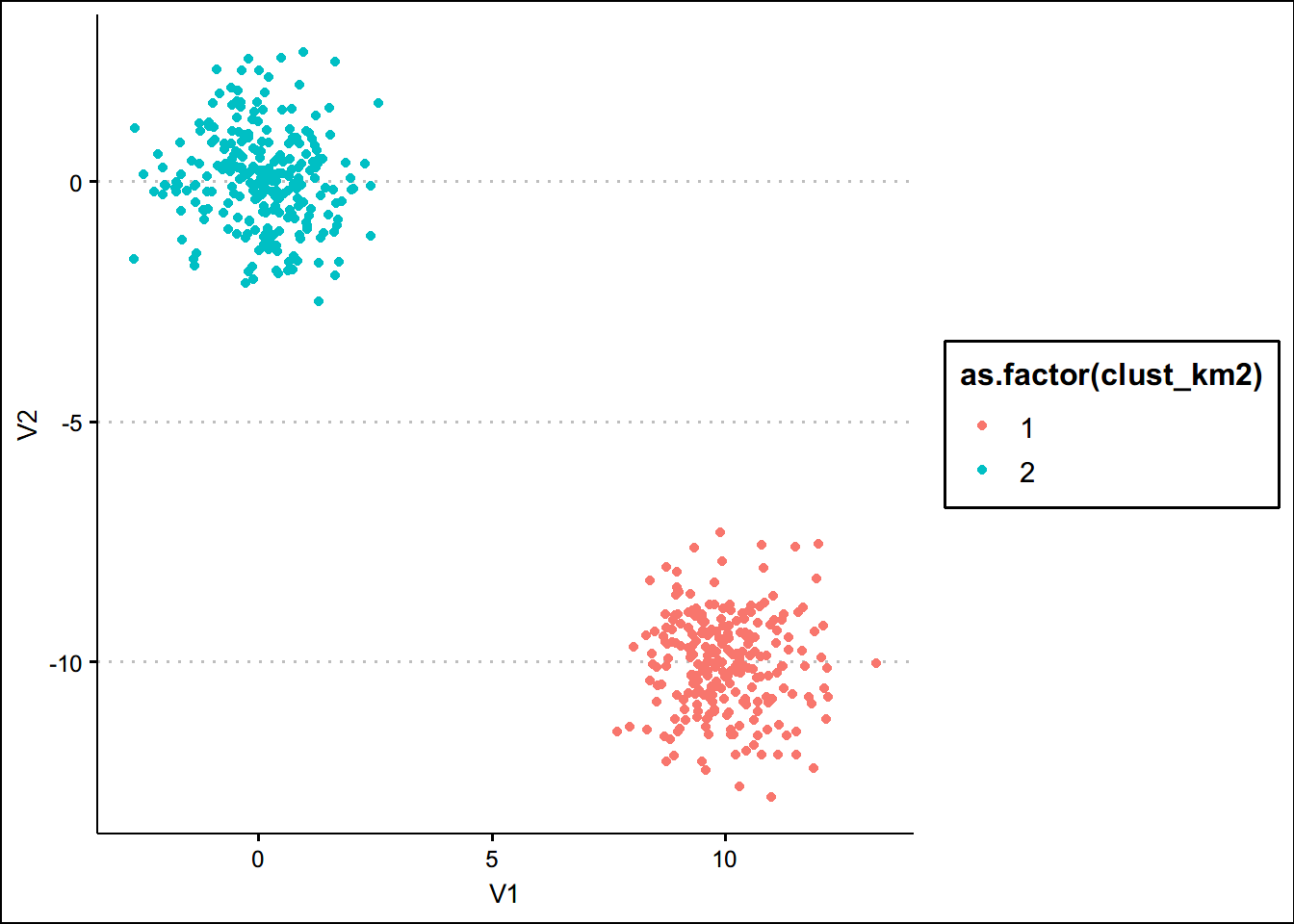
km_out <- kmeans(x,centers = 2)
clust_km2 <-km_out$cluster
x_km2 <- mutate(x,cluster = clust_km2)
#再画一下数据
ggplot(data=x_km2,aes(x=V1,y=V2,color=as.factor(clust_km2)))+geom_point()+theme_clean()如何选择k,如果我们事前不知道k如何处理?
先看个动图
library(haven)
library(factoextra)
aqi <- read_dta("data/data_0224.dta")
aqi <- aqi %>%
select(city,aqi ,pm25,pm10,no2,o3,co,so2) %>%
drop_na()
aqi_sum <- aqi %>%
group_by(city) %>%
summarise(pm25_m =mean(pm25),so2_m = mean(so2),pm10_m=mean(pm10),no2_m=mean(no2),co_m=mean(co))
rownames(aqi_sum) <- aqi_sum$city
aqi_c <- select(aqi_sum, - city)
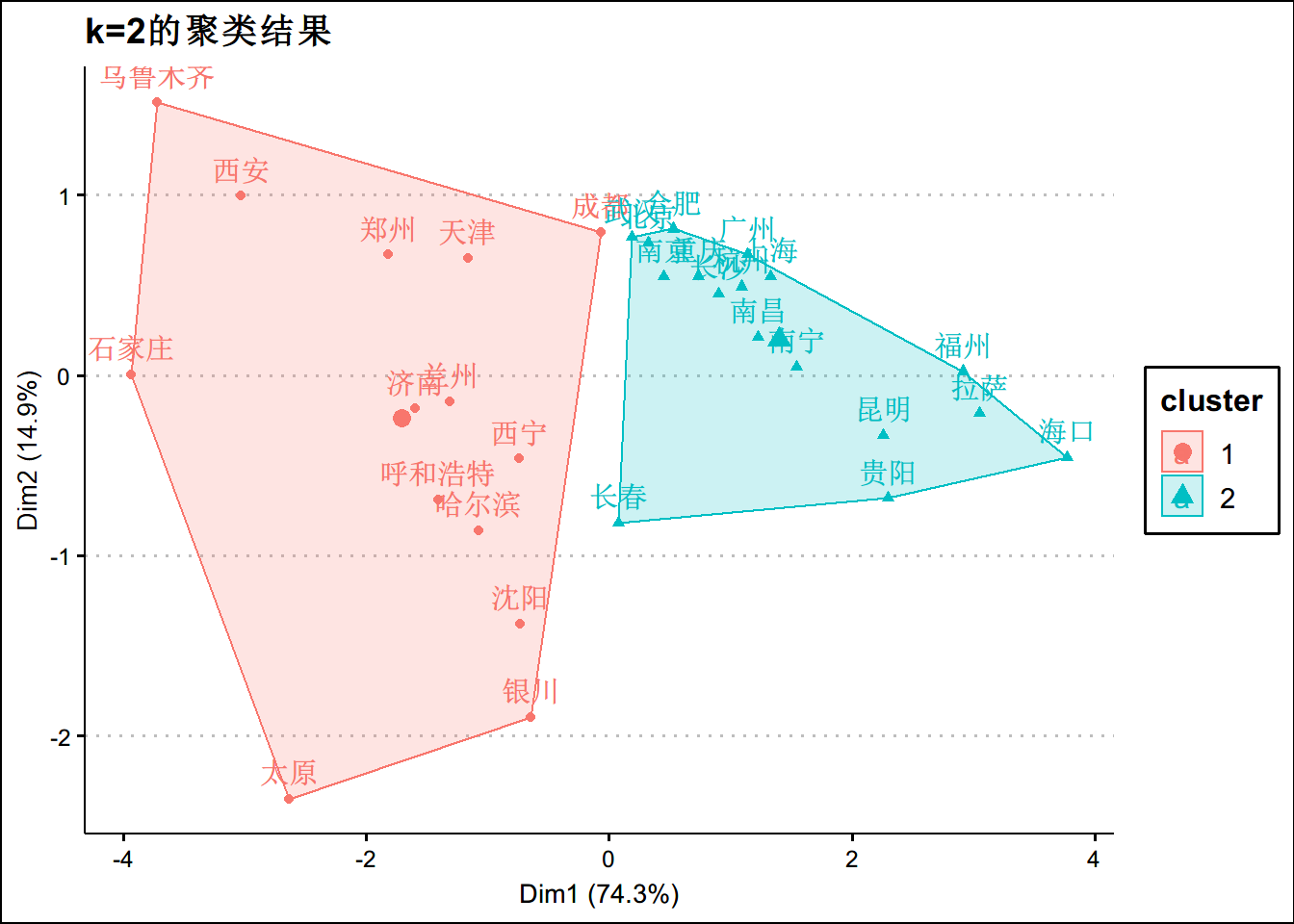
km_out <- kmeans(aqi_c,centers = 2)
fviz_cluster(km_out,data=aqi_c) +theme_clean() + ggtitle("k=2的聚类结果")km_out3 <- kmeans(aqi_c,centers = 3)
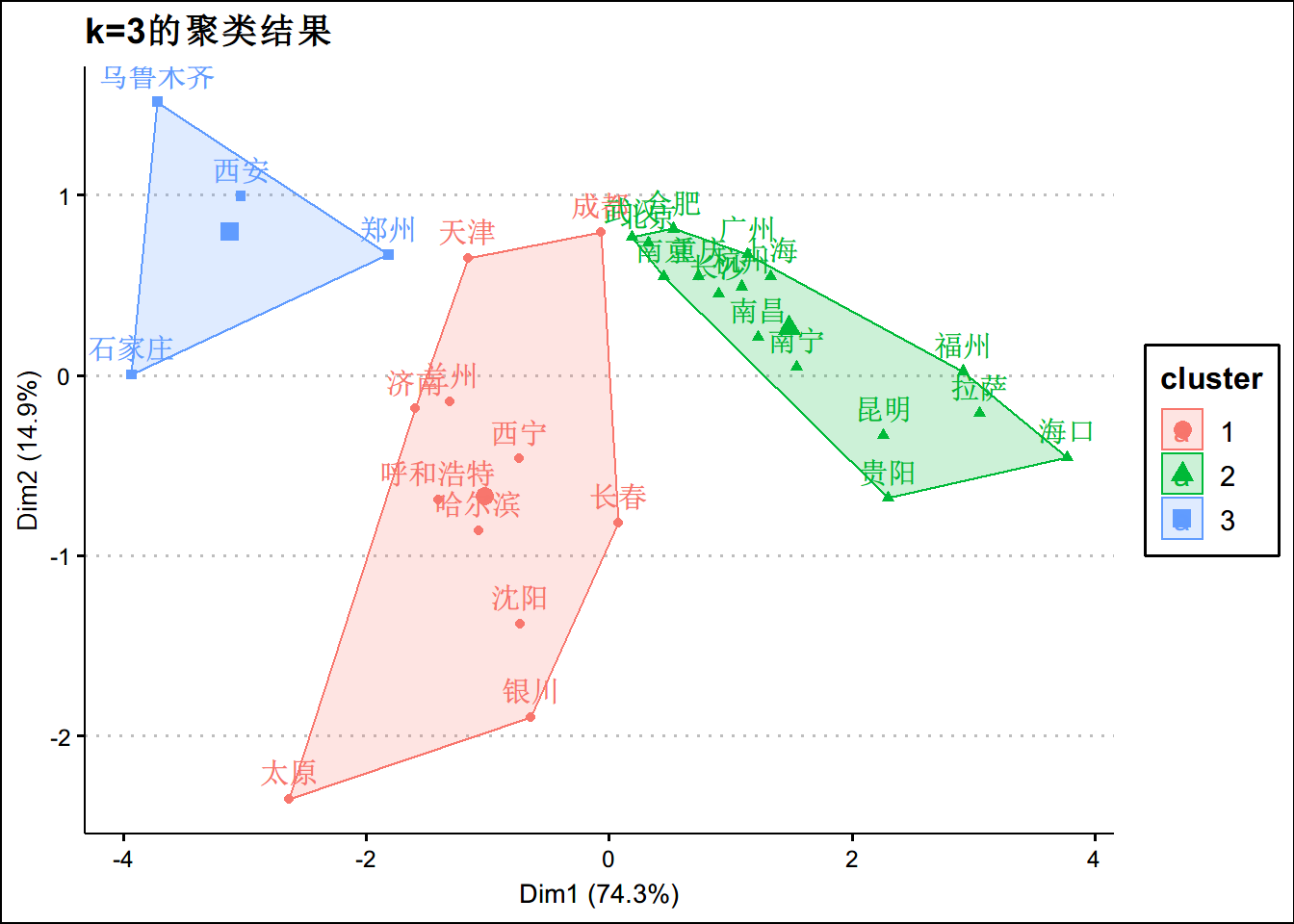
fviz_cluster(km_out3,data=aqi_c) +theme_clean() + ggtitle("k=3的聚类结果")km_out4 <- kmeans(aqi_c,centers = 4)
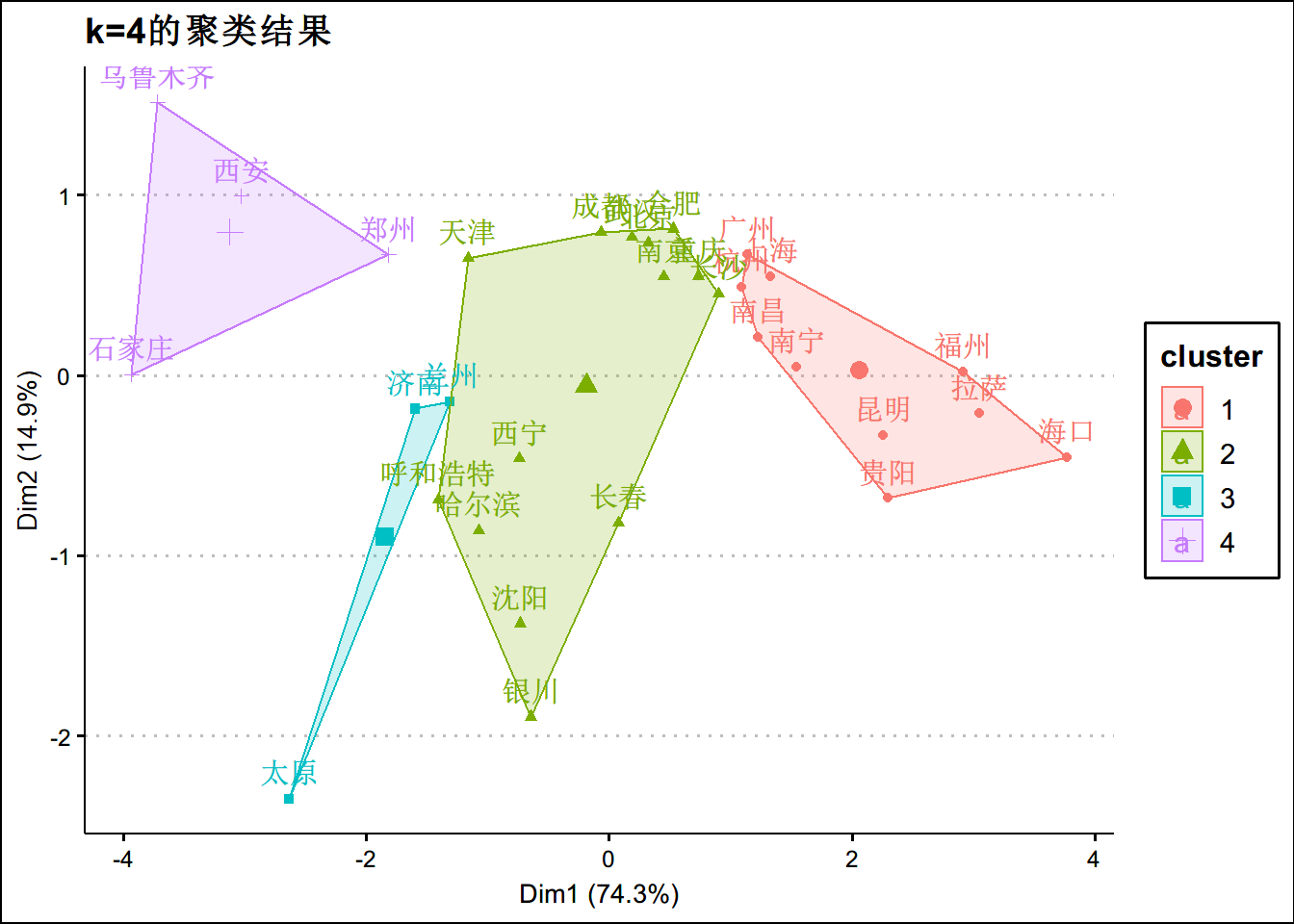
fviz_cluster(km_out4,data=aqi_c) +theme_clean() + ggtitle("k=4的聚类结果")如何寻找最优的k?
Elbow Method
kmean聚类背后的基本思想是定义集群,以使总集群内变化(称为总集群内变化或总集群内平方和)最小化: 是第个集群是集群内的变异。
Elbow Method:
- 针对k的不同值计算kmean。例如,kmean中选取1到10个集群参数
- 对于每个k,计算群集内的总平方和(wss)
- 根据聚类数k绘制wss曲线。
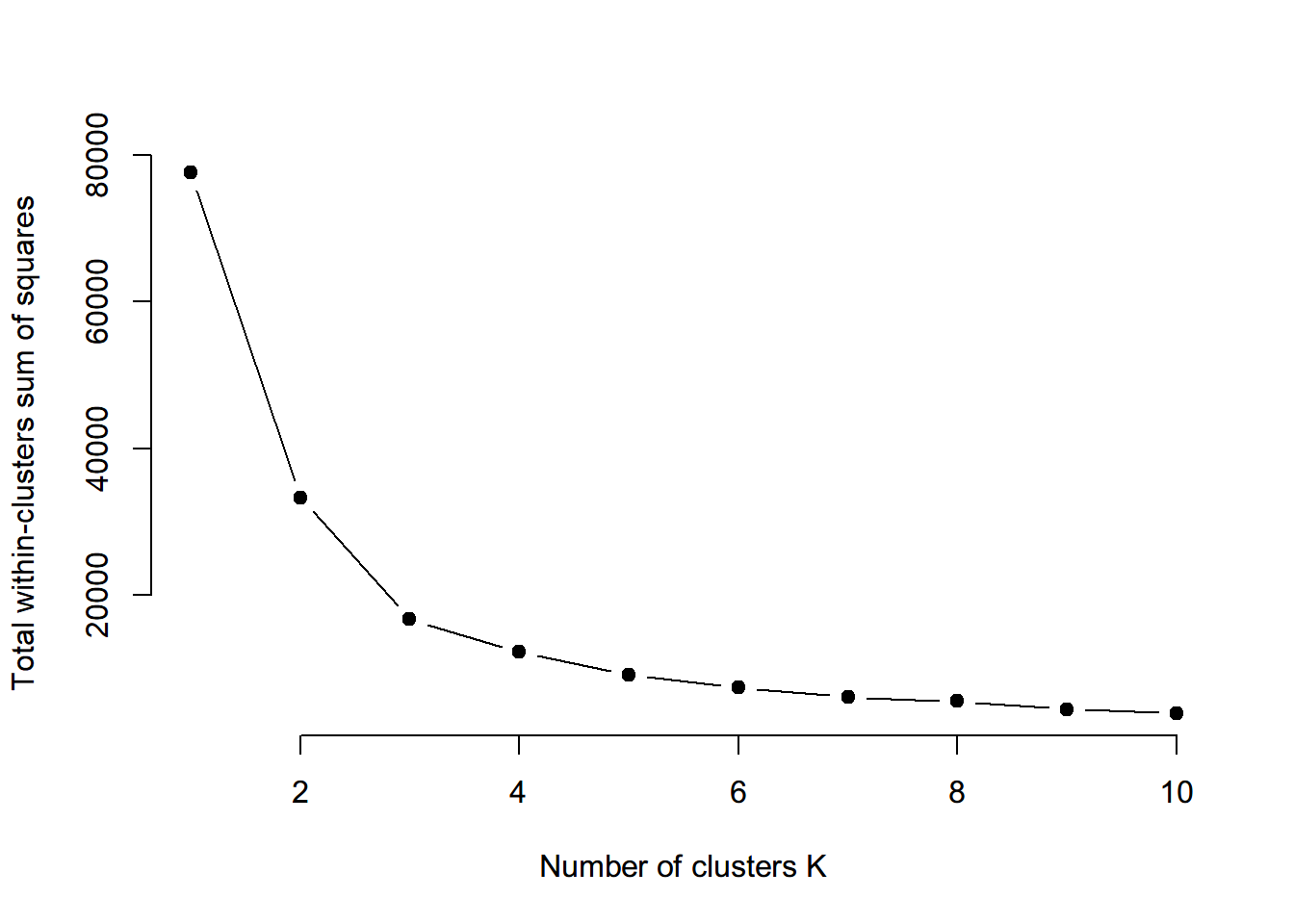
- 曲线中拐点的位置通常被视为适当簇数的指标。 *** 可以通过以下方法实现
set.seed(123)
wss <- function(k) {
kmeans(aqi_c, k, nstart = 10 )$tot.withinss
}
k_val <- 1:10
wss_values <- map_dbl(k_val, wss)
plot(k_val, wss_values,
type="b", pch = 19, frame = FALSE,
xlab="Number of clusters K",
ylab="Total within-clusters sum of squares")#或者直接用便好的函数
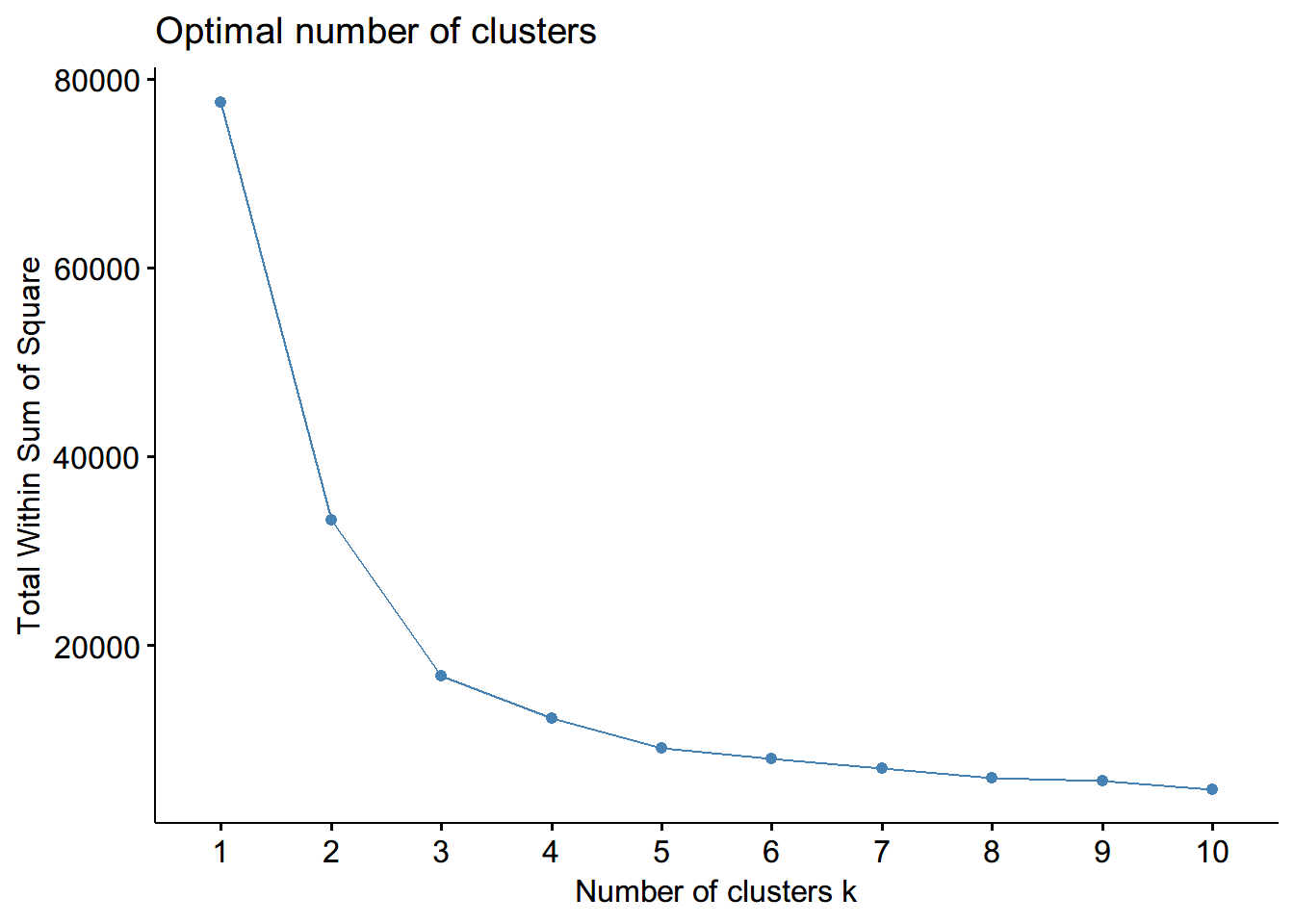
fviz_nbclust(aqi_c, kmeans, method = "wss") #可能选三是合适的